【原文:】
目 录
1 Libsvm下载... 3
2 Libsvm3.0环境变量设置... 3
3 训练和测试数据集下载... 3
4 运行python程序的环境配置... 3
5 LIBSVM 使用的一般步骤是:... 3
6 再来说一下,libsvm-3.0的需要的数据及其格式。... 4
7 Libsvm数据格式制作:... 4
8 Windows版本的工具... 4
9 核函数的使用简介... 8
10 grid.py的使用... 10
11 subset.py使用方法... 11
12 checkdata.py的使用方法... 12
13 easy.py使用方法... 12
14 一个具体的应用实例... 13
15 备注:常见问题的解决办法... 15
1 Libsvm下载
在你给我发的邮件地址 下载libsvm的最新版本,现在最新版本是3.0的,下载地址为
1、 将libsvm-3.0.zip解压,我的解压到了C:\根目录下了。
2 Libsvm3.0环境变量设置
添加环境变量到path:我的电脑->属性->高级->环境变量->系统变量->变量列表里面双击 Path,在变量值最后添加;C:\libsvm-3.0\windows 然后点击确定即可。
3 训练和测试数据集下载
在以下网址 可以下载到用来训练的数据集合用来测试的数据集,这都是一些简单的数据。
4 运行python程序的环境配置
由于有可能使用到C:\libsvm-3.0\tools下的工具,所以的搭建相应的环境,下载两个软件python和gnuplot,下面说一下两个软件的下载地址和配置:
a.对于python,下载地址在上寻找下载,目前版本是3.2的,但是我用的是2.6的,测试成功。安装就默认安装在C:\就行,记得将C:\Python26 添加到系统环境变量中。另外将C:\Python26 中的python.exe添加到C:\libsvm-3.0\windows下面即可。
b.对于gnuplot ,下载地址在 。目前最新版本是4.4.3。他不用安装,直接解压放到C:\根目录即可。(这注:部分不需要了)。
c.下载gp440win32.zip,在网址 下载,这个解压放到C盘根目录,然后拷贝C:\gnuplot\binary中的gnuplot.exe放到C:\libsvm-3.0\windows下面即可。
5 LIBSVM 使用的一般步骤
1)按照LIBSVM软件包所要求的格式准备数据集
2)对数据进行简单的缩放操作;
3)首要考虑选用RBF 核函数;
4)采用交叉验证选择最佳参数C与g ;
5)采用最佳参数C与g 对整个训练集进行训练获取支持向量机模型;
6)利用获取的模型进行测试与预测。
6 libsvm-3.0数据及其格式
该软件使用的训练数据和检验数据文件格式如下:
[label] [index1]:[value1] [index2]:[value2] …
[label] [index1]:[value1] [index2]:[value2] …
Label 就是说class(属于哪一类), 就是你要分类的种类,通常是一些整数。
index 是有順序的索引,通常是连续的整数。就是指特征编号,必须按照升序排列
value 就是特征值,用来 train 的数据,通常是一堆实数组成。
(注:修改训练和测试数据的格式为程序可以识别的程序
目标值 第一维特征编号:第一维特征值 第二维特征编号:第二维特征值…
目标值 第一维特征编号:第一维特征值 第二维特征编号:第二维特征值…
……
目标值 第一维特征编号:第一维特征值 第二维特征编号:第二维特征值…
例如: 2.3 1:5.6 2:3.2
表示训练用的特征有两维,第一维是5.6,第二维是3.2,目标值是2.3
注意:训练和测试数据的格式必须相同,都如上所示。测试数据中的目标值是为了计算误差用 )
7 Libsvm数据格式制作
该过程可以自己使用excel或者编写程序来完成,也可以使用网络上FormatDataLibsvm.xls来完成。 FormatDataLibsvm.xls使用说明:
a. 先将数据按照下列格式存放(注意label放最后面):
value1 value2 … label
value1 value2 … label
…
value1 value2 … label
b. 然后将以上数据粘贴到FormatDataLibsvm.xls中的最左上角单元格,接着工具->宏->执行FormatDataToLibsvm宏。就可以得到libsvm要求的数据格式。测试可用,下载地址在( ),或者自己再google中搜索下载即可。
8 Windows版本的工具
Windows版本的工具svm-scale.exe进行训练和测试数据的归一化,svm-train.exe进行模型训练,svm-predict.exe进行预测,具体步骤和过程在下面介绍。
2、 C:\libsvm-3.0\windows下的工具使用方法
打开C:\libsvm-3.0\windows ,里面有四个windows的exe程序。下面解释一下这四个exe执行程序的意义和用法
①svm-scale.exe: scale输入数据,简单优化数据以提高精度。 扫描数据. 因为原始数据可能范围过大或过小, svmscale可以先将数据重新scale (縮放) 到适当范围使训练与预测速度更快
使用方法:svm-scale.exe [-l lower] [-u upper] [-y y_lower y_upper] [-s save_name] [-r store_name] filename;其中
-l:数据下限标记;
lower:缩放后数据下限 缺省值: lower = -1;
-u:数据上限标记;
upper:缺省值: upper = 1,代表着没有对y进行缩放;
-y:是否对目标值同时进行缩放;
y_lower:为下限值;
y_upper:为上限值;(回归需要对目标进行缩放,因此该参数可以设定为 –y -1 1 );
-s save_name:表示将缩放的规则保存为文件save_name;
-r store_name:表示将缩放规则文件store_name载入后按此缩放;
filename:待缩放的数据文件(要求满足前面所述的格式)。
缩放规则文件可以用文本浏览器打开,看到其格式为:
y
lower upper min max x
lower upper
index1 min1 max1
index2 min2 max2
其中的lower 与upper 与使用时所设置的lower 与upper 含义相同;index 表 示特征序号;min 转换前该特征的最小值;max 转换前该特征的最大值。数据集的缩放结果在此情况下通过DOS窗口输出,当然也可以通过DOS的文件重定向符号“>”将结果另存为指定的文件。该文 件中的参数可用于最后面对目标值的反归一化。反归一化的公式为:
(Value-lower)*(max-min)/(upper - lower)+lower
其中value为归一化后的值,其他参数与前面介绍的相同。
建议将训练数据集与测试数据集放在同一个文本文件中一起归一化,然后再将归一化结果分成训练集和测试集。
② svm-train.exe:训练数据,生成模型
使用方法:svm-train.exe [options] training_set_file [model_file],其中:
options(操作参数):可用的选项即表示的涵义如下所示 -s svm类型:设置SVM 类型,默认值为0,可选类型有(对于回归只能选3或4):
0 – C-SVC
1 – n-SVC
2 -- one-class-SVM
3 – e-SVR
4 – n-SVR
-t 核函数类型:设置核函数类型,默认值为2,可选类型有:
0 -- 线性核:u'*v
1 -- 多项式核: (g*u'*v+ coef 0)deg ree
2 -- RBF 核:e( u v 2) g –
3 -- sigmoid 核:tanh(g*u'*v+ coef 0)
-d degree:核函数中的degree设置,默认值为3;
-g g :设置核函数中的g ,默认值为1/ k ;
-r coef0:设置核函数中的coef 0,默认值为0;
-c cost:设置C- SVC、e - SVR、n - SVR中从惩罚系数C,默认值为1;
-n n :设置n - SVC、one-class-SVM 与n - SVR 中参数n ,默认值0.5;
-p e :设置n - SVR的损失函数中的e ,默认值为0.1;
-m cachesize:设置cache内存大小,以MB为单位,默认值为40;
-e e :设置终止准则中的可容忍偏差,默认值为0.001;
-h shrinking:是否使用启发式,可选值为0 或1,默认值为1;
-b 概率估计:是否计算SVC或SVR的概率估计,可选值0 或1,默认0;
-wi weight:对各类样本的惩罚系数C加权,默认值为1;
-v n:n折交叉验证模式。
training_set_file:是要进行训练的数据集;
model_file:是训练结束后产生的模型文件,该参数如果不设置将采用默认的文件名,也可以设置成自己惯用的文件名。
(注1:其中-g选项中的k是指输入数据中的属性数。操作参数 -v 随机地将数据剖分为n 部分并计算交叉检验准确度和均方根误差。以上这些参数设置可以按照SVM 的类型和核函数所支持的参数进行任意组合,如果设置的参数在函数或SVM 类型中没有也不会产生影响,程序不会接受该参数;如果应有的参数设置不正确,参数将采用默认值。)
(注2:网上实验中的参数-s取3,-t取2(默认)还需确定的参数是-c,-g,-p
另, 实验中所需调整的重要参数是-c 和 –g,-c和-g的调整除了自己根据经验试之外,还可以使用gridregression.py对这两个参数进行优化。(需要补充)
该优化过程需要用到Python(测试是2.5),Gnuplot(测试是4.2),gridregression.py(1、该文件需要修改路径2、下载地址在,再试没有使用过)。然后在命令行下面运行:
python.exe gridregression.py -log2c -10,10,1 -log2g -10,10,1 -log2p -10,10,1 -s 3 –t 2 -v 5 -svmtrain E:\libsvm\libsvm-2.86\windows\svm-train.exe -gnuplot E:\libsvm\libsvm-2.86\gnuplot\bin\pgnuplot.exe E:\libsvm\libsvm-2.86\windows\train.txt > gridregression_feature.parameter
以上三个路径根据实际安装情况进行修改。
-log2c:是给出参数c的范围和步长
-log2g:是给出参数g的范围和步长
-log2p:是给出参数p的范围和步长上面三个参数可以用默认范围和步长
-s:选择SVM类型,也是只能选3或者4 -t是选择核函数
-v :10 将训练数据分成10份做交叉验证。默认为5
为了方便将gridregression.py可以将其存放在python.exe安装目录下
trian.txt为训练数据,参数存放在gridregression_feature.parameter中,可以自己命名。
搜索结束后可以在gridregression_feature.parameter中最后一行看到最优参数。
其中,最后一行的第一个参数即为-c,第二个为-g,第三个为-p,最后一个参数为均方误差。前三个参数可以直接用于模型的训练。 然后,根据搜索得到的参数,重新训练,得到模型。
例如:svmtrain.exe -s 3 -p 0.0001 -t 2 -g 32 -c 0.53125 -n 0.99 feature.scaled ;
再例如:运行svmtrain.exe heart_scale会得出下面结果输出:
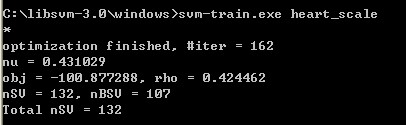
其中,
#iter为迭代次数,
nu 是你选择的核函数类型的参数,
obj为SVM文件转换为的二次规划求解得到的最小值,
rho为判决函数的偏置项b,
nSV 为标准支持向量个数(0<a[i]<c),
nBSV为边界上的支持向量个数(a[i]=c),
Total nSV为支持向量总个数(对于两类来说,因为只有一个分类模型Total nSV = nSV,但是对于多类,这个是各个分类模型的nSV之和)。
训练后的模型保存为文件heart_scale.model,用记事本打开其内容如下:
svm_type c_svc % 训练所采用的svm类型,此处为C- SVC
kernel_type rbf %训练采用的核函数类型,此处为RBF核
gamma 0.0769231 %设置核函数中的g ,默认值为1/ k
nr_class 2 %分类时的类别数,此处为两分类问题
total_sv 132 %总共的支持向量个数
rho 0.424462 %决策函数中的常数项b
label 1 -1 %类别标签
nr_sv 64 68 %各类别标签对应的支持向量个数
SV %以下为支持向量
1 1:0.166667 2:1 3:-0.333333 4:-0.433962 5:-0.383562 6:-1 7:-1 8:0.0687023 9:-1 10:-0.903226 11:-1 12:-1 13:1
0.5104832128985164 1:0.125 2:1 3:0.333333 4:-0.320755 5:-0.406393 6:1 7:1 8:0.0839695 9:1 10:-0.806452 12:-0.333333 13:0.5
1 1:0.333333 2:1 3:-1 4:-0.245283 5:-0.506849 6:-1 7:-1 8:0.129771 9:-1 10:-0.16129 12:0.333333 13:-1
1 1:0.208333 2:1 3:0.333333 4:-0.660377 5:-0.525114 6:-1 7:1 8:0.435115 9:-1 10:-0.193548 12:-0.333333 13:1
③ svm-predict.exe:用来测试训练结果的准确率
使用方法:svm-predict.exe [options] test_file model_file output_file
options(操作参数):
-b probability_estimates:是否需要进行概率估计预测,可选值为0或1,默认值为0。
model_file:是由svm-train.exe 产生的模型文件;
test_file:是要进行预测的数据文件;
output_file:是svmpredict 的输出文件,表示预测的结果值。
(注:输出结果包括均方误差(Mean squared error)和相关系数(Squared correlation coefficient)。)
例如:svmpredict.exe feature_test.scaled feature.scaled.model feature_test.predicted
其中feature_test.scaled是归一化后的测试特征文件名,feature.scaled.model是训练好的模型,SVM预测的值在feature_test.predicted中 。
9 核函数的使用简介
(1)核函数发展历史
早在1964年Aizermann等在势函数方法的研究中就将该技术引入到机器学习领域,但是直到1992年Vapnik等利用该技术成功地将线性 SVMs推广到非线性SVMs时其潜力才得以充分挖掘。而核函数的理论则更为古老,Mercer定理可以追溯到1909年,再生核希尔伯特空间 (ReproducingKernel Hilbert Space, RKHS)研究是在20世纪40年代开始的。
(2)核函数方法原理
根据模式识别理论,低维空间线性不可分的模式通过非线性映射到高维特征空间则可能实现线性可分,但是如果直接采用这种技术在高维空间进行分类或回归,则存 在确定非线性映射函数的形式和参数、特征空间维数等问题,而最大的障碍则是在高维特征空间运算时存在的“维数灾难”。采用核函数技术可以有效地解决这样问 题。
设x,z∈X,X属于R(n)空间,非线性函数Φ实现输入间X到特征空间F的映射,其中F属于R(m),n<<m。根据核函数技术有:
K(x,z) =<Φ(x),Φ(z) > (1)
其中:<, >为内积,K(x,z)为核函数。从式(1)可以看出,核函数将m维高维空间的内积运算转化为n维低维输入空间的核函数计算,从而巧妙地解决了在高 维特征空间中计算的“维数灾难”等问题,从而为在高维特征空间解决复杂的分类或回归问题奠定了理论基础。
(3)核函数特点
核函数方法的广泛应用,与其特点是分不开的:
1)核函数的引入避免了“维数灾难”,大大减小了计算量。而输入空间的维数n对核函数矩阵无影响,因此,核函数方法可以有效处理高维输入。
2)无需知道非线性变换函数Φ的形式和参数.
3)核函数的形式和参数的变化会隐式地改变从输入空间到特征空间的映射,进而对特征空间的性质产生影响,最终改变各种核函数方法的性能。
4)核函数方法可以和不同的算法相结合,形成多种不同的基于核函数技术的方法,且这两部分的设计可以单独进行,并可以为不同的应用选择不同的核函数和算法。
(4)常见核函数
核函数的确定并不困难,满足Mercer定理的函数都可以作为核函数。常用的核函数可分为两类,即内积核函数和平移不变核函数,如:
1)高斯核函数K(x,xi) =exp(-||x-xi||2/2σ2;
2)多项式核函数K(x,xi)=(x·xi+1)^d, d=1,2,…,N;
3)感知器核函数K(x,xi) =tanh(βxi+b);
4)样条核函数K(x,xi) = B2n+1(x-xi)。
(5)核函数方法实施步骤
核函数方法是一种模块化(Modularity)方法,它可分为核函数设计和算法设计两个部分,具体为:
1)收集和整理样本,并进行标准化;
2)选择或构造核函数;
3)用核函数将样本变换成为核函数矩阵,这一步相当于将输入数据通过非线性函数映射到高维 特征空间;
4)在特征空间对核函数矩阵实施各种线性算法;
5)得到输入空间中的非线性模型。
显然,将样本数据核化成核函数矩阵是核函数方法中的关键。注意到核函数矩阵是l×l的对称矩阵,其中l为样本数。
(6)核函数在模式识别中的应用
1)新方法。主要用在基于结构风险最小化(Structural Risk Minimization,SRM)的SVM中。
2)传统方法改造。如核主元分析(kernel PCA)、核主元回归(kernel PCR)、核部分最小二乘法(kernel PLS)、核Fisher判别分析(Kernel Fisher Discriminator, KFD)、核独立主元分析(Kernel Independent Component Analysis,KICA)等,这些方法在模式识别等不同领域的应用中都表现了很好的性能。
10 grid.py使用方法
文件grid.py是对C-SVC的参数c和γ做优选的,原理也是网格遍历,假设我们要对目录C:\libsvm-3.0下的样本文件heart_scale做优选,其具体用法为:
第一步:打开C:\libsvm-3.0下的tools文件夹,找到grid.py文件,将其拷贝到C:\libsvm-3.0\windows下。用python打开(不能双击,而要右键选择“Edit with IDLE”),修改svmtrain_exe和gnuplot_exe的路径。
svmtrain_exe = r"C:\libsvm-3.0\windows\svm-train.exe"
gnuplot_exe = r"C:\gnuplot\binary\pgnuplot.exe"
(注:这里面还有一个是对非win32的,可以不用改,只改#example for windows下的两行就可以)
第二步:运行cmd,进入dos环境,定位到C:\libsvm-3.0\windows文件夹,这里是放置grid.py的地方。
第三步:输入以下命令:
python grid.py heart_scale
(注:还要把数据文件heart_scale放置到C:\libsvm-3.0\windows下面啊)
你就会看到dos窗口中飞速乱串的[local]数据,以及一个gnuplot的动态绘图窗口。大约过10秒钟,就会停止。Dos窗口中的[local]数据时局部最优值,这个不用管,直接看最后一行:
2048.0 0.0001220703125 84.0741
其意义表示:C = 2048.0;γ=0.0001220703125(γ是哪个参数?参看LibSVM学习(三)中svmtrain的参数说明);交叉验证精度CV Rate = 84.0741%,这就是最优结果。
第四步:打开目录C:\libsvm-3.0\windows,我们可以看到新生成了两个文件:heart_scale.out和heart_scale.png,第一个文件就是搜索过程中的[local]和最优数据,第二文件就是gnuplot图像。
现在,grid.py已经运行完了,你可以把最优参数输入到svm-train.exe中进行训练了。当然了,你在当中某一步很可能出现问题,下面就需要注意的问题说明一下:
1)grid.py和svm-train的版本要统一,也就是说你不能用2.6的grid.py去调用2.89的svm-train。
2)你的目录中如果有空格,比如C:\program files\ libsvm\...,那么无论是在第一步还是第二步,在运行cmd后,请把目录改成C:\program files\ libsvm\...
3) 第三步的命令问题。首先要看你定位到哪个目录,那么其下的文件就不需要带路径,否则就要带。像我们上面的命令,我当前的目录是C:\libsvm-3.0\windows,那么其下的grid.py和heart_scale等文件就不需要加路径,如果python.exe是在C:\python26\下,不在当前目录下,那么就要要加路径。其命令就可以改成:
C:\python26\python grid.py heart_scale
总起来说,命令为python 目标文件 样本文件,其原则是要让系统找得到文件。假如系统提示你“不是内部或外部命令”,说明你python的路径错误,而如果是‘not found file’的提示,很可能是其他两个文件路径错误。
4)假如,你仍旧出现问题,那么请换一下python或者gnuplot的版本,目前python最新版本是3.1,但是好像会出问题,老一点的版本2.4或2.5的兼容性会更好。
11 subset.py使用方法
训练大的数据集会是非常耗时的事情,在某一些场合上,我们可以先工作在一个更小的一个训练集的子集上实验,这也就是subset.py的用处所在,它的主要功能就是从大的数据中抽取一定数量的数据子集,它的应用格式是如下的:
subset.py [options] dataset number [output1] [output2]
options:
-s method : method of selection (default 0)
0 -- stratified selection (classification only)
1 -- random selection
dataset : 数据文件
number: 要选定的subset的个数
output1: the subset(optional)
output2: the rest of data(optional)
例如: python subset.py heart_scale 100 file1 file2
From heart_scale 100 samples are randomly selected and stored in
file1. All remaining instances are stored in file2.
(注:具体参见C:\libsvm-3.0\tools下的README,使用记事本或者写字板打开)
12 checkdata.py使用方法
checkdata.py 用于检测样本集存储格式是否正确在控制台下定位到subset.py所在的目录(将其拷贝到C:\libsvm-3.0\windows下)运行:
python checkdata.py heart_scale
运行结果:

No error. (表示数据文件heart_scale格式没有错误,可以进行后续的调用)
13 easy.py使用方法
文件easy.py对样本文件做了“一条龙服务”,从参数优选,到文件预测。因此,其对grid.py、svm-train.exe、svm-scal.exe和svm-predict.exe都进行了调用(当然还有必须的python和gnuplot)。因此,运行easy.py需要保证这些文件的路径都要正确。当然还需要样本文件和预测文件,再简单的测试中,可以使用heart_scale作为训练样本,预测文件同样使用heart_scale,只是我们复制一份后将其改名为heart_test,下面说一下使用方法:
第一步:将easy.py拷贝到C:\libsvm-3.0\windows目录下,用python打开(不能双击,而要右键选择“Edit with IDLE”),修改# example for windows下的几个路径:
svmscale_exe = r"C:\libsvm-3.0\windows\svm-scale.exe"
svmtrain_exe = r"C:\libsvm-3.0\windows\svm-train.exe"
svmpredict_exe = r"C:\libsvm-3.0\windows\svm-predict.exe"
gnuplot_exe = r"C:\gnuplot\binary \pgnuplot.exe"
grid_py = r"C:\libsvm-3.0\windows\grid.py"
第二步:运行cmd,进入dos环境,定位到放置easy.py的目录C:\libsvm-3.0\windows。
第三步:输入命令:
python easy.py heart_scale heart_test
你就会看到一个gnuplot的动态绘图窗口。大约20s以后停止,dos窗口显示为:
Scaling training data...
Cross validation...
Best c=2048.0, g=0.0001220703125 CV rate=84.0741
Training...
Output model: heart_scale.model
Scaling testing data...
Testing...
Accuracy = 85.1852% (230/270) (classification)
Output prediction: heart_test.predict
这就是最终预测结果,可以看到第三行就是调用grid.py的结果。在C:\libsvm-3.0\windows下你会看到又多了7个文件,都是以前我们碰到的过程文件,都可以用记事本打开。
14 具体的应用实例
1) 以libsvm-3.0中的heart_scale作为训练数据和测试数据,同时已经将python2.6安装至c盘,并将grid.py文件中关于gnuplot路径的默认值修改为实际解压缩后的路径,将heart_scale、grid.py和python.exe拷贝至C:\libsvm-3.0\windows文件夹下。
2)运行cmd,定位到目录C:\libsvm-3.0\windows如下所示:
3)输入如下命令
svm-train.exe heart_scale
得到结果图:

optimization finished, #iter = 162
nu = 0.431029
obj = -100.877288, rho = 0.424462
nSV = 132, nBSV = 107
Total nSV = 132
(注:参数的意义上面有介绍)
4)此时,已经得到heart_scale.model,进行预测:
svm-predict heart_scale heart_scale.model heart_scale.out

Accuracy = 86.6667% (234/270) (classification)
正确率为Accuracy = 86.6667%。
5)进行参数最优估计,运行命令
python grid.py heart_scale
大概运行10s的样子,命令如下所示
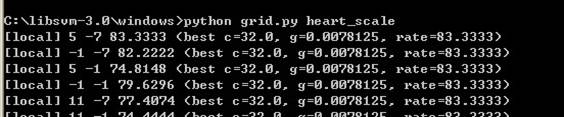
运行到最后得到

注意到最后一行的参数了吧,就是最有参数:
最优参数c=2048,g=0.0001220703125.
6)然后在调用命令
svm-train.exe -c 2048 -g 0.0001220703125 heart_scale得到model

7)调用命令:
svm-predict.exe heart_scale heart_scale.model heart_scale.out
得到结果窗口如下:
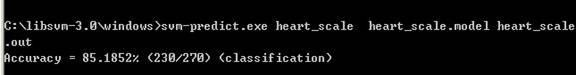
得到的正确率为Accuracy = 85.1852%.这块还有点迷惑?为什么正确率降低了?当然也可以结合subset.py 和 easy.py 实现自动化过程。详见上面的相关介绍
15 备注
常见问题的解决办法
网址为:
1)
Scaling training data...
Cross validation...
Traceback (most recent call last):
File "easy.py", line 61, in ?
c,g,rate = map(float,last_line.split())
ValueError: need more than 0 values to unpack
[解析] 说明你的grid.py运行出现错误,你可以参照上部分“grid.py使用方法”运行一下就会发现问题。另外,有的说是相对路径的问题,建议找到easy.py的以下部分:
cmd = "%s -svmtrain %s -gnuplot %s %s" % (grid_py, svmtrain_exe, gnuplot_exe, scaled_file)
改成
cmd = "%s %s -svmtrain %s -gnuplot %s %s" % (python_path, grid_py, svmtrain_exe, gnuplot_exe, scaled_file)
2)
Traceback (most recent call last)
File "grid.py", line 349, in ?
main()
File "grid.py", line 344, in main
redraw(db)
File "grid.py", line 132, in redraw
gnuplot.write("set term windows\n")
IOError [Errno 22] Invalid argument
[解析] 说明你的gnuplot.exe在调用过程中出现问题,要么是你的路径不对,要么是你的版本不对,请检查。
3)
Traceback (most recent call last):
File "C:\Python24\lib\threading.py", line 442, in __bootstrap
self.run()
File "c:\libsvm\tools\gridregression.py", line 212, in run
self.job_queue.put((cexp,gexp,pexp))
File "C:\Python24\lib\Queue.py", line 88, in put
self._put(item)
File "c:\libsvm\tools\gridregression.py", line 268, in _put
self.queue.insert(0,item)
AttributeError: 'collections.deque' object has no attribute 'insert
[解析] 很显然,你调用的是gridregression.py,其是用来做回归用的。如果你调用easy.py也出现这种问题按照原作者的说法,这里是因为你的python调用出现错误,很可能是版本不对,如果是2.4的版本,请把easy.py中的
self.queue.insert(0,item)
改成
if sys.hexversion >= 0x020400A1:
self.queue.appendleft(item)
else
self.queue.insert(0,item)